RIP技术原理及相关产品
发布日期:2012-12-10 浏览次数:5599
RIP技术原理及相关产品
95%的人类基因组并不编码基因,而是产生大量的非编码RNA,真正编码蛋白质的基因只占人类总基因组的约2%。这些非编码RNA在生长发育的各个阶段都发挥着重要的调节作用,与艾滋病、白血病、糖尿病、畸形等多种病变密切相关,并且参与着干细胞和表观遗传学调控。RNA结合蛋白(RBPs)通过细胞核或细胞质中的RNA识别基序(RRM)或者RNA结合区(RBD)来结合RNA,这种结合作用取决于RBP的类型和所结合的RNA序列。而RNA-蛋白复合物驱动了几乎所有细胞过程的基因表达的转录后调控,包括剪接(splicing)、出核转运(nuclear export)、mRNA 稳定性以及蛋白转译过程。因此,对基因调控的了解就有赖于确定这些过程中RNA的结合的变化,RNA研究也被越来越多的科学家重视。RIP技术逐渐成为RNA研究领域的一项常规方法,特别是转录后调控网络;同样也是研究肿瘤发生和转移中microRNA失调的有力工具。
RIP技术(RNA Binding Protein Immunoprecipitation,RNA结合蛋白免疫沉淀),是研究细胞内RNA与蛋白结合情况的技术,是了解转录后调控网络动态过程的有力工具,能帮助发现miRNA的调节靶点。RIP技术运用针对目标蛋白的抗体把相应的RNA-蛋白复合物沉淀下来,然后经过分离纯化就可以对结合在复合物上的RNA进行分析。RIP是染色质免疫沉淀ChIP技术的类似应用,但由于研究对象是RNA-蛋白复合物而不是DNA-蛋白复合物,RIP实验的优化条件与ChIP实验不太相同(如复合物不需要固定,RIP反应体系中的试剂和抗体绝对不能含有RNA酶,抗体需经RIP实验验证等)。RIP技术下游结合microarray技术被称为RIP-Chip,更高通量地了解癌症以及其它疾病整体水平的RNA变化。
RIP用抗体或表位标记物捕获细胞核内或细胞质中内源性的RNA结合蛋白,RNA结合蛋白及其结合的RNA一起分离出来,结合的RNA序列再通过microarray(RIP-Chip),定量RT-PCR或高通量测序(RIP-Seq)方法来鉴定 。
首先对细胞进行裂解;在裂解液中加入抗体及蛋白A/G磁珠,接着用磁极固定磁珠复合物,并洗脱未结合的蛋白-RNA复合物;提取蛋白-RNA中的RNA,纯化后进行下游实验,如qRT-PCR,微阵列(RIP-Chip)、高通量测序(RIP-Seq)等。
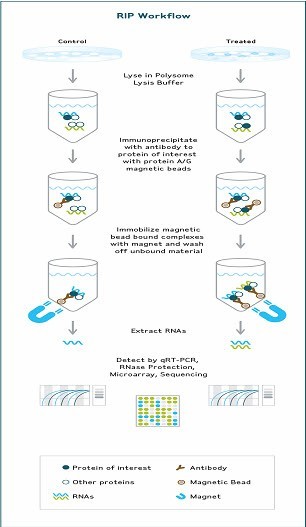 |
RIP的一大优势在于,通过MS(质谱技术),RIP可以同时鉴定RNPs中的蛋白组分和其相关联的调控蛋白。环境条件的改变或者在细胞分化过程中,RNP复合物会发生重塑,而RNA mapping和MS技术可以对这种重塑进行检测。
作为表观遗传学研究的领军者,默克密理博(Merck Millipore)总结了其在研究DNA与蛋白相互作用的经验,推出了用于研究RNA与蛋白相互作用的试剂盒(RIP™试剂盒、目录号:17-700、17-701)以及其他一系列相关产品,为该研究领域的飞速发展提供了便利的条件。
- (一)、新型EZ-RIP™磁珠试剂盒:
- 1.方便,快速,易于操作。
- 2.包括了所有试剂,All-in-one!
- 3.提供阴性和阳性对照抗体,对照引物对,特别适用于该技术的初次使用者。
- 4.提供优化的磁珠,适于沉淀RNA-蛋白复合物,效率高,非特异性背景低。
- 5.高品质,稳定性好。
- 6.结果重复率高。
- (二)、多种RIP相关抗体
【上一篇】ChIP实验原理及步骤
【下一篇】BD PMG协助发现调控T细胞分化增殖的关键因素


